
한의학 연구에 활용되는 네트워크 약리학 데이터베이스들의 특성 비교 : 대표적 시나리오 중심으로
Ⓒ The Society of Pathology in Korean Medicine, The Physiological Society of Korean Medicine
Abstract
Network pharmacology is a research field based on system biology by combining computational analysis with molecular biological experiments to discover drug targets and molecular biological mechanisms. Therefore, a variety of Traditional Chinese Medicine(TCM) databases and tools are developed and used in Korean medicine research study. In this review, we conducted a comparative study of characteristics of TCM databases, focusing on several representative network pharmacology scenarios. We selected 10 TCM related databases from Database Commons and Pubmed based on their publication year and citation numbers. We analyzed source of data, characteristics, service components such as analytical functions of them. Also, we introduced several representative scenarios for exploring the efficacy of herb or herbal formula based on papers related to system biology. Our findings suggest that researchers can use network pharmacology based scenarios to explore the therapeutic mechanisms and targets between disease and herb or herbal formula. In conclusion, this review aims to provide researchers with ideas for selecting appropriate databases for Korean medicine research.
Keywords:
Network pharmacology, Korean medicine, Herbal formula, Scenarios서 론
네트워크 약리학은 시스템 생물학, 유전체학, 단백질체학 등을 기반으로 하는 연구 분야로 in vivo와 in vitro 실험을 통해 구축된 대량의 데이터들을 전산분석기법 및 분자생물학적 실험과 통합하여 약물의 타겟과 분자생물학적 메커니즘 등을 발견하는 연구 분야이다1). 네트워크 약리학은 2007년 Andrew L. Hopkins에 의해 처음 제시되었는데 2007년 이전에는 약물의 one-target, one-disease와 같이 각 요소들을 분해하여 질병에 대한 타겟을 환원주의 방법으로 예측하였으나 최근에는 약물의 단일 타겟이 아닌 다중 타겟을 기반으로 한 multi-compound, multi-target, multi-disease 의 방법으로 전환되고 있다2).
한약은 군신좌사의 이론에 의한 다양한 약재로 조합되어 있으며 수 많은 성분들을 포함하고 있다. 이러한 다양한 약재들의 조합은 단일 약재의 치료 효능을 높이거나 독성과 부작용을 완화시켜 궁극적으로 보다 높은 치료 효과를 기대할 수 있게 하는 장점이 있다3). 그러나 이러한 한약의 다중성분으로 인한 특성은 단일 성분의 타겟을 확인하는 생물학적 효능 실험을 적용하기에는 어려움을 낳게 한다4). 이러한 특성으로 인해 최근에는 한약의 효능을 분석하기 위한 네트워크 약리학 연구가 많이 이루어지고 있으며 이를 통해 다양한 성분과 타겟들 간의 상호작용을 밝히고 생물학적 시스템 수준에서의 기전을 규명하고자 하는 시도가 이루어지고 있다5).
최근에는 이러한 노력의 일환으로 여러 네트워크 약리학 데이터베이스 및 도구들이 개발되고 있다. 대표적으로는 농축 분석(enrichment analysis) 기능을 제공하는 BATMAN-TCM, TCM-ID, Symmap 그리고 HERB 등이 있고 약동학적 특징을 갖는 TCMSP, TM-MC 2.0 등이 있으며 각각 데이터베이스만의 장점을 보유하고 있다. 이에 대해 Zhang 등6)의 연구에서는 네트워크 약리학 연구에 활용될 수 있는 HIT, TCMID, TCMSP 그리고 TCM-Mesh 그리고 TCM database@Taiwan등의 데이터베이스들 및 도구들의 특징을 비교하고 한약의 네트워크 약리학 연구 사례들을 소개하였다. Xue 등7)의 연구에서는 TCMSP, TCMID, ETCM, BATMAN-TCM 등의 한의학 데이터베이스들의 특징과 장점 및 보완할 점에 대해 정리하였다. Zhao 등8)의 연구에서는 한약에 따라 사용한 데이터베이스의 종류 및 생물학적 기전에 대해 서술하였으며 TCMSP, ETCM, BATMAN-TCM 그리고 HIT 과 같은 데이터베이스들의 특징과 분석할 수 있는 내용에 대해 정리하여 소개한바 있다.
국내 연구에서는 Beck 등4)의 논문에서 한약의 효능 분석에 사용될 수 있는 TCM-ID, TCMID, TCMSP, HIT 등의 데이터베이스의 특징들과 한약의 효능을 분석한 네트워크 약리학 연구사례들을 정리하였다. 그리고 Lee 등9)의 논문에서는 TCMSP, TCMID 와 Symmap 등의 데이터베이스들의 설명과 한약-성분-타겟에 대한 정보를 제공하는지 살펴보았다. 이외에도 Hwang 등10)의 논문에서는 네트워크 약리학 연구에 활용 가능한 TCM-ID, TCMDatabase@Taiwan, TCMID과 같은 한약 관련 데이터베이스들의 특징을 비교하였다.
이와 같이 국내외에서 한의학 관련 데이터베이스 연구가 다수 이루어지고 있다. 그러나 실제 이러한 데이터를 이용해서 연구를 하고자 하는 연구자의 입장에서 각 데이터베이스별 특징과 제공하는 기능의 차이를 비교하는 연구는 이루어진 바 없다. 이에 본 연구에서는 대표적인 네트워크 약리학 분석 시나리오에 따라 각 데이터베이스의 특성 및 제공하는 정보를 비교하였다. 더불어 연구 목적별로 보다 적합한 데이터베이스를 선택하는데 참고가 될 수 있도록 자료를 도출하고자 하였다.
연구대상 및 방법
1. 분석 대상 데이터베이스 선정
한약 효능 연구에 사용할 수 있는 데이터베이스를 선정하기 위해 생물학적 데이터베이스가 정리된 Database Commons (https://ngdc.cncb.ac.cn/databasecommons/) 에서 z score(citation of database/database age)이 5 이상인 데이터베이스들을 선정하였다. 그리고 선정한 각 데이터베이스의 이름을 키워드로 Pubmed에서 검색하여 2011년부터 2023년 까지 네트워크 약리학 연구에 사용된 논문의 수를 조사하여 다빈도 순으로 정리하였다. 그 외 신규로 발표되어 아직 활용도가 낮은 데이터베이스의 경우는 연구자 2인의 합의를 거쳐 추가 하였다.
2. 데이터베이스의 특징 비교
각 데이터베이스에서 제공하는 서비스를 홈페이지에 기술된 사용법과 해당 데이터베이스를 활용하여 발표된 논문을 참고하여 데이터베이스가 포함하고 있는 데이터와 분석 기능의 특징을 비교하여 표로 정리하였다. 데이터는 한약, 성분, 약동학적 특성, 타겟 그리고 질환 또는 변증 관련 정보의 포함 여부를 살펴보았고 분석 기능으로는 농축 분석(enrichment analysis)과 네트워크 시각화(network visualization)기능을 제공하는지 비교하였다. 이와 더불어 한의학 연구자가 각 데이터베이스들을 사용하고자 할 때 대표적 시나리오를 중심으로 장점을 살펴보았다.
시스템 약리학의 한의학적 활용 및 한의학 데이터베이스들을 리뷰한 Zhang 등6)이 제시한 연구 유형을 기본으로 하고 2011년 부터 2023년 까지 Pubmed에서 ‘systems pharmacology’, ‘network pharmacology TCM’ 또는 ‘system biology’의 키워드로 검색한 결과를 분류하여 3개의 대표적인 연구 유형으로 정리하였다. 각 유형별로 대표적인 활용 사례를20-24) 참고하여 실제 연구자의 연구 진행 방향을 고려해 분석 단계를 순서도 형식으로 표현하였다.
결 과
1. 한의 관련 대표적 시스템 약리학 데이터베이스
한약의 효능 분석에 사용되어지는 다양한 데이터베이스들 중 연구에 활용된 논문의 수와 구축된 시기를 고려하여 비교적 자주 사용되는 시스템을 인용 횟수 순으로 Table 1에 나타내었다. 그리고 각 데이터베이스들의 특징과 제공하는 정보 리스트에 대한 출처를 기술하였다. 100회 이상 인용된 데이터베이스는 TCMSP, BATMAN-TCM, TCMID 로 TCMSP 데이터베이스는 2012년에 구축된 시스템이며 이를 활용한 네트워크 약리학적 효능 연구는 1,230건으로 조사되어 활발히 수행되고 있었으며 BATMAN-TCM은 2016년에 구축 된 후 2023년 까지 155건이 인용되었다. TCMID는 2013년에 발표 된 이후 현재까지 115건의 한약 효능 연구에 활용됨을 확인하였다. 이외에도 2024년 2월까지 발표된 최신 데이터베이스들 중 ETCM, Symmap, HERB, TCM-ID, HIT, DCABM-TCM 그리고 TM-MC의 경우 위의 데이터베이스에서 제공하지 않는 각 데이터베이스만의 고유한 특징 또는 장점을 보유하고 있어 최종 분석에 포함하였다.
TCMSP는 흡수, 분포, 대사 및 배설의 약동학적 성질(ADME)을 제공하므로 약재의 활성 성분의 선별이 가능하며 중국 약전으로부터 약재의 정보를 참고하였다. BATMAN-TCM 2.0은 약재 또는 처방 단위의 분석이 가능하며 cut-off score과 p-value를 통해 타겟을 예측할 수 있다. TCMID 2.0은 한약과 현대 약물에 대한 정보 뿐만 아니라 약재 및 약재의 성분에 대한 질량 분석(mass spectrometry) 실험 데이터를 제공한다. ETCM 2.0은 처방과 약재와 더불어 중국 특허 약물에 대한 정보를 제공하는 것이 특징이다. 또한 약물 유사도 및 독성 평가와 같은 약리학적 특징을 약물 개발에 적용할 수 있다. Symmap 데이터베이스는 한의학적 증상과 현대 의학의 증상을 연결한 네트워크를 제공한다. 최근 개발된 HERB 2.0 데이터베이스는 RNA sequencing 과 microarray의 전사체 분석을 기반으로한 고처리량 실험(high-throughput experiments) 데이터를 제공하며Connectivity Map (CMap) 데이터베이스와 연동되어 한약과 현대 약물 간의 유사성에 대한 정보를 제공한다.
이외에도 한약 효능 연구에 다수 활용되어지고 있는 TCM-ID은 한약재의 성분 분자 구조와 기능적인 특성을 제공하는 종합적인 데이터베이스이다. HIT 2.0의 경우 텍스트 마이닝 방법을 사용하여 약재에 대한 성분 정보를 원문과 연결하여 제공한다. DCABM-TCM 은 실험 결과로부터 한약재 성분이 인체에 그대로 흡수된 물질인prototype과 대사된 물질인 metabolite의 성분에 대한 정보를 제공하는 것이 특징이다. TM-MC 2.0은 한국, 중국 그리고 일본의 약전에서 약재의 정보를 참고하였으며 크로마토그래피 실험 논문을 참고하여 약재의 성분을 전문가가 수동으로 검수하고 타데이터베이스를 참고하지 않고 독자적으로 기 발표된 논문만을 참고로 데이터베이스를 구축함에 따라 정보의 신뢰성이 높다는 점이 특징이다.
2. 한약 효능 분석에 사용되는 대표적인 네트워크 약리학 연구 시나리오들
대표적인 시스템 약리학적 연구 사례들은 3가지가 있다. 첫번째 유형은Wu등20)의 연구에서와 같이 네트워크 약리학 데이터베이스를 활용하여 1) 한약재의 성분 중 약동학적 특성을 고려한 주요 성분을 도출하고 2) 특정 목표 질환과 관련된 유전자 타겟을 선정하여 성분-타겟-질환 네트워크 기반의 농축 분석을 통해 3) 기존에 알려진 한약재나 처방이 목표 질환에 효과를 나타내는 생물학적 기전을 이해하기 위한 유형으로 이해할 수 있다(Fig. 1A).
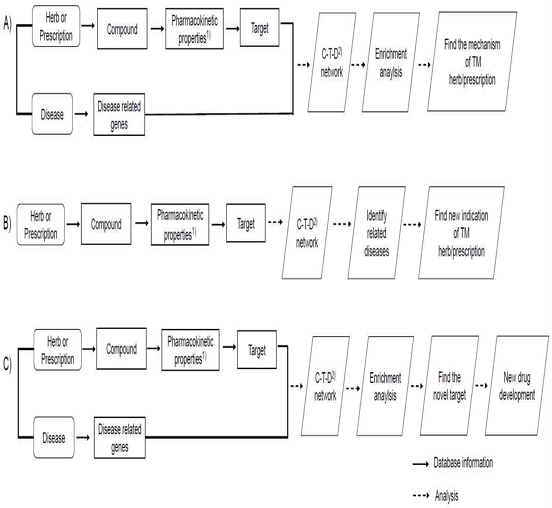
Flowchart of Traditional Medicine(TM) research scenarios based on network pharmacology method. 1) Pharmacokinetic properties are optional in TM research. 2) C-T-D network : Compound-Target-Disease network
두번째 유형은 Li 등21)과 Tao 등22)의 연구에서와 같이 1) 사전에 목표하는 질환을 정하지 않은 상태로 시스템 약리학 데이터베이스를 통해 한약재의 성분 중 약동학적 특성을 고려한 주요 성분을 도출하고 2) 해당 성분과 관련된 유전자 타겟을 선정하여 3) 성분-타겟-질환 네트워크 분석을 통해 한약이 활용될 수 있는 후보 질환 및 새로운 적응증을 탐색하는 연구로 이해할 수 있다(Fig. 1B).
세번째는 Zhang 등23,24)의 연구에서와 같이 1) 한약재 또는 처방의 효과가 있다고 알려진 질환을 중심으로 2) 관련된 한약재(들)와 질환사이에 성분-타겟-질환 네트워크 기반의 농축 분석을 통해 3) 질환과 관련된 한약의 핵심 기전을 분석함으로써 기존의 현대의학에서 고려하지 못한 새로운 기전에 기반한 신약 개발 후보를 탐색하는 연구로 이해할 수 있다(Fig. 1C).
위와 같이 한약재와 질환이 공유하는 치료기전을 찾아내거나, 한약재를 중심으로 다양한 질환 타겟을 연구하여 새로운 현대의학적 적응증을 탐색하거나, 질환을 중심으로 공통 타겟 기전을 탐색하여 새로운 신약개발 방법론을 탐색하는 등의 3가지 대표적인 연구 유형과 해당 연구의 대표적 시나리오를 통합하여 하나의 도표로 나타내면 Fig. 2와 같이 하나의 통합된 순서도 형식으로 표현 할 수 있다.
3. 네트워크 약리학 시나리오에 따른 데이터베이스 특징
위의 과정을 통해 3가지 시나리오를 통합한 순서도를 중심으로 선정된 데이터베이스의 특징을 분석하여 Table 2에 제시하였다. Disease/TCM Syndrome 열에서는 해당 데이터베이스에 수재된 현대적 질환, 또는 한의학적 변증 등과 관련된 정보의 유무와 그 데이터의 양을 한눈에 비교할 수 있도록 하였으며, Disease related genes열에서는 해당 데이터베이스에서 자체적으로 제공하는 질환과 연관된 유전자 정보제공 유무를 정리하였다. Herbal formula열에서는 단일 한약재 뿐 아니라 처방과 관련된 정보제공 유무 및 제공하는 처방의 개수를 정리하였으며, Herb열에서는 한약재와 관련된 정보와 제공하는 한약재 관련 정보의 개수를 정리하였다. Compound 열에서는 한약재내에 포함된 성분 정보의 유무와 개수를 정리하였다. Pharmacokinetic properties열에서는 각 한약재의 약동학적 특성과 관련된 정보 제공유무를 정리하였으며 Target열에서는 타겟 유전자와 관련된 정보제공 유무 및 그 데이터의 양을 정리하였다. Pathway열에서는 타겟과 관련된 생물학적 기전 정보 제공 유무를 표시하였으며 다른 서비스와 연계하여 제공하는 경우는 해당 서비스를 표시하였다. Enrichment analysis열에서는 농축 분석 기능의 여부를 표시하였으며 자체적으로 제공하는 경우가 아니라 외부 서비스와 연계되는 경우는 연계되는 서비스를 표시하였다. Network Visualization에서는 네트워크 약리학에서 자주 사용되는 시각화 도구의 제공 여부를 표시하였다.

Comparison of TM related databases. The comparison contains data components, analytical functions and general statistics of each component
아래 도표에 따르면 가장 많이 사용되고 있는 데이터베이스인 TCMSP는 단독으로 사용시 분석 기능이 없어 타데이터베이스 또는 분석 서비스와 결합하여 사용해야함을 알 수 있다. 그러나 흡수, 분포, 대사 및 배설과 같은 약동학적 특성을 통해서 활성 성분을 선별할 수 있는 장점이 있으므로 약동학적 특성에 중점을 두고 분석을 하고자 하는 경우 유용하게 활용할 수 있음을 알 수 있다. 또한 두번째로 많은 논문이 출간되고 있는BATMAN-TCM 2.0의 경우에는 TCMSP가 제공하지 않는 처방에 대한 정보를 제공함은 물론 모든 영역에서 서비스 및 데이터를 제공하여 연구의 전 과정을 하나의 데이터베이스에서 처리할 수 있다는 장점이 있다. 이와 더불어, 한의학적 변증과 질환에 대한 정보를 모두 제공하는 데이터베이스로는 TCMID 2.0, Symmap 그리고 ETCM 2.0 이 있으며 Symmap은 한의학적 증상과 관련된 현대의학적 증상, 타겟 및 질환 정보를 제공하는 특징이 있다. 이와 같이 각 데이터베이스별로 한약재, 관련성분, 타겟 메커니즘 등의 데이터 제공 수준에 차이가 있어 이러한 특징들을 비교하여 연구자의 목적에 맞게 데이터베이스를 선택할 필요가 있다.
Table 2에서 제공된 데이터베이스의 특징 중 약동학적 특징을 제공하는 데이터베이스들에 대한 정보를 Table 3에서 보다 상세하게 분류하였다. 약동학적 특징 분석은 약물의 생리학적 및 생화학적 작용과 약물이 인체에 일으키는 반응을 연구할 수 있다. 자주 사용되는 지표들은 Ru 등11)에 따르면 OB, HL, DL, FASA, Caco-2 permeability, TPSA, BBB 그리고 Lipinski’s rule of 5등이 있다. 이중 대표적인 특징인 약물 생용성(Oral Bioavailability)은 약물을 경구 투여시 체내로 흡수되는 약물의 양을 비율로 나타낸 지표로 한약재 내에 포함된 성분이라 할지라도 소화흡수 과정에서 얼마나 실제로 흡수되어 활용되는지를 평가할 수 있는 지표이며 약물 유사도(Drug Likeness)는 한약재 내에 포함된 성분의 분자구조 특성이 알려진 약물과 얼마나 유사한지를 평가함으로써 실제 약물로써 효과가 있을지를 추정하는데 사용된다. 또한 약물 유사성의 지표로 분자량, 소수성, 수소 결합의 donor와 acceptor를 고려하는 리핀스키 규칙(Lipinski’s rule of 5)등 역시 한약재 내의 약물이 약동학적으로 얼마나 실제 인체 내에서 약성을 나타낼 수 있는지를 추정하는 방법 중 하나이다. Table 3에 제공되는 정보를 통해 연구자의 목적에 맞는 약동학적 정보를 제공하는 DB를 선택하거나 또는 다른 DB와 조합하여 활용할 필요가 있다.
고 찰
한약은 천연물을 사용하는 특성상 다중 성분- 다중 타겟의 특징을 갖고 있어 그동안 약효의 예측에 어려움을 겪어 왔다. 그러나 최근 시스템 생물학의 발달로 단일 표적 기반이 아닌 복잡한 다중 표적을 가지는 한약재의 효능 및 관련 기전을 빠른 시간 안에 분석할 수 있게 되었다.
예를 들어 Wu 등20)은 이러한 시스템 생물학의 도구를 이용하여 한약재의 치료 효과와 관련된 생물학적 기전을 탐색 하였으며 그 결과 갈근의 genistein, puerain, beta-sitosterol 등의 성분이 항염증 및 항산화 기전을 통해 고혈압 개선에 효과를 나타낼 수 있을 것이라고 보고하였다. Liu 등21)은 한약재가 가지고 있는 잠재적 목표 질환을 시스템 생물학을 통해 탐색 하였으며 그 결과 감초가 호흡기 질환, 심혈관 질환 뿐만 아니라 과민성 대장 증후군 및 구토 증상과 같은 장 질환 등의 다양한 치료를 나타낼 수 있을 것이라고 보고하였다. 또한 단일 약재가 아닌 여러 약재가 혼합된 처방에서도 이러한 시스템 생물학적인 접근은 복잡한 약물의 상호작용을 예측하여 그 효과 기전이나 목표 질환 등을 예측할 수 있는데 대표적인 사례로 Li 등25)은 황련해독탕의 quercetin, kaempferol, wogonin 그리고 baicalein 등의 성분들이 세포 사멸, 혈관 신생 등과 같은 생물학적 기능을 조절하는 것으로 예측된다고 보고하였으며 Tao 등22)은 다양한 시스템 생물학 데이터베이스들을 활용하여 울금, 치자, 사향, 용뇌로 구성된 한의처방이 심혈관 질환 뿐만 아니라 종양, 신경계 질환에도 치료 효과가 있을 것이라고 예측하였다. 또한 Fang 등26)의 연구에서는 목표 질환을 치료할 수 있는 새로운 약재 조합을 찾아내기 위해 텍스트 마이닝(text-mining) 기법을 활용하여 기존의 다양한 연구 문헌을 통해 알츠하이머 질환 치료 효능이 있는 울금, 인삼, 단삼 등의 약재를 선정하고 한약 데이터베이스들을 활용하여 신경 활성 리간드-수용체 상호작용, 대사 경로, 칼슘 신호 전달 경로 등의 기전들 과의 관련성을 통해 알츠하이머 질환에 치료 효능을 예측하여 보고하였다.
이처럼 네트워크 약리학 방법을 통해 한약의 효능을 예측할 수 있을 뿐만 아니라 한의학의 진단과 치료 과정의 기반이 되는 변증에 대한 생물학적 기전을 규명하고자 하는 연구 또한 수행되고 있다. Wang 등27)은 脾氣虛證(비양허증)의 증상에 해당되는 만성 소화기 질환과 관련된 단백질 및 생물학적 기전을 파악함으로써 한의학의 변증을 네트워크 약리학을 통해 이해하고자 하였으며 Tran 등28)은 기허증(氣虛證)에 사용되는 한약재의 고유한 타겟 메커니즘을 분석하여 기허의 생물학적 기전을 파악하고자 하는 등 단순한 질환 치료 뿐만 아니라 해당 한약재 또는 적응증과 관련된 생물학적 기전에 대한 이해를 기반으로 한의학적 변증 개념에 대한 이해까지 확장하는 연구도 추진되고 있다.
이러한 연구를 수행하기 위해서는 각 한약재에 함유된 성분에 대한 정보는 물론 각 성분의 분자 구조 및 약동학적 특성, 관련된 유전자 및 생물학적 메커니즘 등에 대한 다양한 정보가 필요하다. 이를 제공하는 것이 네트워크 약리학 데이터베이스들로 한약재와 관련된 네트워크 약리학 데이터베이스는 대표적으로 TCMSP, BATMAN-TCM, TCM-ID 등이 존재한다. 이러한 데이터베이스들은 각각 특성에 따라 제공하는 서비스에 차이를 보인다. TCMSP의 경우에는 경구 투여 시 약물 활성과 관련된 정보를 제공하여 활용할 수 있도록 하고 있으나 자체 분석 도구는 제공하고 있지 않으며 BATMAN-TCM의 경우는 자체 알고리즘을 통해 one-stop으로 타겟 질환이나 메커니즘을 탐색할 수 있는 서비스를 제공한다. 최근 이러한 한약 데이터베이스들을 비교하는 연구가 활발히 이루어지고 있는데 그 중 Hwang 등10)의 연구에서는 다양한 시스템 생물학 데이터베이스들을 Li 등29)의 연구에 따라 약물 관련 데이터베이스, 생체 분자 및 표현형 네트워크 리소스로 나누어 각 데이터베이스들의 특징을 비교하였다. 그리고 시스템 생물학 도구들 중 성분 뿐만 아니라 타겟 및 질병과의 관련성을 통합적으로 제공하는 TCM-ID, TCMDatabase@Taiwan, TCMGeneDIT, TCMID, TCMSP 그리고 HIT 데이터베이스들을 비교하였다. 또한 TCMSP 데이터베이스를 활용한 한약의 효능 연구 사례를 감초의 예시를 통해 분석하였으며 사상체질의학 약물과 TCMSP 데이터베이스가 포함하는 약물의 상호 비교를 통해 한의학 데이터베이스를 설계 시 고려해야할 사항을 도출하였다. Beck 등4)의 연구에서는 Ru 등11)의 연구에 따라 자주 사용되고 있는 한약 데이터베이스들인 TCM-ID, TCMDatabase@Taiwan, TCMGeneDIT, TCMID 등의 특징을 비교하고 2012년부터 2015년 까지 발표된 시스템 약리학을 기반으로 한 약물 효능 연구 사례들을 소개하였다. Fan 등30)의 논문에서는 약재, 성분, 타겟 뿐만 아니라 표현형 및 변증 관련 데이터베이스들의 특징과 분석 기능을 비교하였다. Lee 등31)의 연구에서는 약재, 성분, 타겟 그리고 질병 관련 데이터베이스들로 나누어 특징을 비교하였다. 그러나 실제 네트워크 약리학을 이용하여 한약재 또는 한약 처방과 관련된 질환 및 메커니즘을 탐색하고자 하는 연구자들에게 위와 같은 기능적 분류만으로는 실제 연구자가 어떤 데이터베이스를 사용하는 것이 가장 편리하고 적절한지를 판단하기 어렵다는 단점이 있다. 이에 본 연구에서는 한약의 효능 연구에 활용되어지는 대표적인 시나리오들을 살펴보고 이를 기반으로 시스템 생물학 데이터베이스들의 특징 및 기능을 분석해보고자 하였다.
본 연구에서는 비교적 한의학 연구에 자주 사용되어지는 데이터베이스들은 물론 최근에 발표되거나 업데이트 된 BATMAN-TCM 2.0, TM-MC 2.0, HERB 2.0 등과 DCABM-TCM 데이터베이스 등을 분석 대상에 포함시켰다. 뿐만 아니라, 대표적인 연구 사례를 유형 별로 나누어 순서도 형식으로 시나리오를 제시함으로써 연구자들이 향후에 한약 데이터베이스들을 목적과 연구 방법에 맞게 참고할 수 있도록 하였다. BATMAN-TCM 2.0과 HERB 2.0, ETCM 2.0, DCABM-TCM 과 같은 데이터베이스는 세가지 시나리오에 모두 대응되는 서비스를 갖추고 있어 하나의 데이터베이스 내에서 다양한 예비 분석을 할 수 있는 장점을 갖추고 있다. TCMSP 나 TM-MC 2.0 등은 한약재가 가지고 있는 성분의 약동학적 특성을 다수 제공하여 실제 경구 투여 시 약리 활성을 예측하는데 중점을 두는 연구자가 활용하기에 유용한 정보를 제공한다.
그러나 이러한 장점 외에 각 데이터베이스가 제공하는 정보의 특성에 따른 단점도 존재한다. 예를 들어 TM-MC 2.0의 경우는 한국, 중국 일본 약전을 모두 포함하고 있으나 그 외의 TCMSP 등과 같이 약재 정보의 출처가 중국 약전으로 국한된 경우는 단순히 한약재 명만으로 검색하였을 경우 해당 한약재가 한국의 기원식물과 다른 경우가 있을 수 있어 약재 검색 시 주의가 필요하다. 羌活(강활), 芥子(개자), 그리고 藿香(곽향) 등과 같은 약재들의 기원종의 경우 한국과 중국 약전에 따라 각기 다르며 성분 조성 및 약리 효능 등이 달라질 수 있어 반드시 이를 고려한 분석이 필요하다32). 또한 데이터베이스의 구축 시 기존 데이터베이스를 활용하는 과정에서 오류가 확산될 수 있는 우려가 존재한다. HIT 2.0과 TM-MC 2.0은 타데이터베이스를 참고하지 않고 독자적인 근거문서에 기반하여 성분-타겟 DB를 구축하였으며 관련된 근거를 제공하여 확인할 수 있도록 하고 있어 한약재-성분 데이터의 정확성이 중요한 연구자들이 참고하기에 우수한 장점을 보유하고 있다. 그러나 그 외의 데이터베이스는 기존 데이터베이스를 인용한 후 추가로 보완하는 형태로 구축되어 기존 오류가 반복되어 나타날 수 있는 우려가 있다.
또한 한약 효능 예측 연구에 있어서 네트워크 약리학 방법은 실제 한약의 기전을 온전히 설명하기에는 부족한 점이 많다. 약재의 구성성분이 충분히 알려지지 않은 한약의 경우는 활용이 어려우며, 또한 연구가 많이 되고 있는 질환에 비해 연구가 부족한 질환의 경우도 관련된 유전자나 생물학적 기전이 알려져 있지 않아 활용이 불가능하다. 또한 성분-표적 예측 방식은 단순히 성분과 표적 사이의 상호작용을 알려줌으로써 가능성을 예측하는데 필요한 정보를 제공하는 한정적인 역할을 한다. 이로 인해 최근에는 단순한 시스템 생물학적 예측 외에 실제 동물 실험이나 세포 실험 등을 통해 확인된 결과만이 인정받는 추세로 나아가고 있다.
그러나 비록 단순한 예측이라 할지라도 기존에 너무 복잡해서 접근할 수 없었던 한약의 효과에 대한 이해를 도모할 수 있다는 점에서 한의학적인 활용에서의 가치는 여전히 높다고 할 수 있다. 다만, 본 연구에서는 각 시스템에서 공식적으로 제공하는 정보를 중심으로 분석을 수행함에 따라 실제 한약재 또는 처방과 질환을 비교하여 대입하면서 각 데이터베이스의 장단점과 결과의 차이 등을 사례중심으로 분석하지는 못하였다. 또한 다양한 사용자의 연구 목적을 모두 고려하기에는 그 가능성이 너무 다양함에 따라 대표적으로 가장 많이 검색되는 3가지 연구 유형을 중심으로 제한적으로 분석하였다는 단점이 있다. 또한 각 데이터베이스들이 제공하고 있는 데이터의 신뢰도를 비교 평가하거나 성분-타겟 추정을 위한 알고리즘의 우수성을 비교 평가하지는 못하였다. 이러한 점을 고려할 때 본 연구에서 제시한 분석 외에 제공되지 않은 여러가지 장단점이 실제 연구 과정에서 드러날 수 있다는 한계가 있다. 그러나 이러한 한계를 고려하여 잘 활용한다면 본 연구에서 제시된 시나리오 중심의 데이터베이스 특성 비교 결과는 향후 한약의 타겟 질환 탐색이나 치료 기전을 예측 하고자 하는 연구자들에게 보다 적절한 도구를 선택하는데 필요한 시간을 줄이거나 최적의 연구계획을 수립하는데 도움이 될 수 있을 것이다.
결 론
한의학 연구에 자주 사용되는 대표적 시나리오를 중심으로 네트워크약리학 데이터베이스들의 특징을 비교한 결과 각 데이터베이스별로 제공하는 데이터의 양과 종류, 분석 기능 등이 서비스별로 상당한 차이가 있음을 확인하였으며, 연구 목적에 맞는 시나리오에 따라 적절한 데이터베이스를 활용할 경우 보다 효과적인 연구에 도움이 될 수 있을 것이다.
Acknowledgments
본 연구는 한국한의학연구원 주요사업을 통해 연구비의 지원을 받아 수행되었습니다. (과제고유번호 : KSN1923111)
References
-
Zhao L, Zhang H, Li N, Chen J, Xu H, Wang Y, et al. Network pharmacology, a promising approach to reveal the pharmacology mechanism of Chinese medicine formula. J Ethnopharmacol. 2023;309:116306.
[https://doi.org/10.1016/j.jep.2023.116306]

-
Hopkins AL. Network pharmacology. Nat Biotechnol. 2007;25(10):1110-1.
[https://doi.org/10.1038/nbt1007-1110]

-
Pan Z, Li M, Jin Z, Sun D, Zhang D, Hu B, et al. Research status of Chinese medicine formula based on network pharmacology. Pharmacological Research Modern Chinese Medicine. 2022;4:100-32.
[https://doi.org/10.1016/j.jep.2023.116306]

-
Beck JM, Seo HK, Kwon YK. Review of Network Pharmacological Approaches on Korean Medicine. Vol 30, Journal of Physiology & amp; Pathology in Korean Medicine. Korean Society of Oriental Pathology; 2016. p. 419.
[https://doi.org/10.15188/kjopp.2016.12.30.6.419]

-
Yang HY, Liu ML, Luo P, Yao XS, Zhou H. Network pharmacology provides a systematic approach to understanding the treatment of ischemic heart diseases with traditional Chinese medicine. Phytomedicine. 2022;104:154268.
[https://doi.org/10.1016/j.phymed.2022.154268]

-
Zhang R, Zhu X, Bai H, Ning K. Network Pharmacology Databases for Traditional Chinese Medicine: Review and Assessment. Front Pharmacol. 2019;10:123.
[https://doi.org/10.3389/fphar.2019.00123.eCollection2019]

-
Xue T, Gong S, Xie T-H, Li W-J, Huang J-P. Development status and trend of traditional Chinese medicine database. 2022.
[https://doi.org/10.53388/MHM2022B0825001]

-
Zhao L, Zhang H, Li N, Chen J, Xu H, Wang Y, et al. Network pharmacology, a promising approach to reveal the pharmacology mechanism of Chinese medicine formula. J Ethnopharmacol. 2023;309:116306.
[https://doi.org/10.1016/j.jep.2023.116306]

-
Lee WY, Kim CE, Lee CY. A Novel Method to investigating Korean Medicine Theory : Drug-centered Approach Employing Network Pharmacology. Korean Society of Oriental Pathology; 2021.
[https://doi.org/10.15188/kjopp.2021.10.35.5.125]

-
Hwang SM, Beck J, Suh SY, Kwon YK. Status of Construction of TCM Network Pharmacology Databases and Potential Application of TCMSP to Korean Traditional Medicine-mainly with Sasang-related Herbs. Journal of Physiology & Pathology in Korean Medicine. 2015;29(6):443-50.
[https://doi.org/10.15188/kjopp.2015.12.12.29.6.443]

-
Ru J, Li P, Wang J, Zhou W, Li B, Huang C, et al. TCMSP: a database of systems pharmacology for drug discovery from herbal medicines. J Cheminform. 2014;6:13.
[https://doi.org/10.1186/1758-2946-6-13]

-
Kong X, Liu C, Zhang Z, Cheng M, Mei Z, Li X, et al. BATMAN-TCM 2.0: an enhanced integrative database for known and predicted interactions between traditional Chinese medicine ingredients and target proteins. Nucleic Acids Res. 2024;52(D1):D1110-d20.
[https://doi.org/10.1093/nar/gkad926]

-
Huang L, Xie D, Yu Y, Liu H, Shi Y, Shi T, et al. TCMID 2.0: a comprehensive resource for TCM. Nucleic Acids Res. 2018;46(D1):D1117-d20.
[https://doi.org/10.1093/nar/gkx1028]

-
Zhang Y, Li X, Shi Y, Chen T, Xu Z, Wang P, et al. ETCM v2.0: An update with comprehensive resource and rich annotations for traditional Chinese medicine. Acta Pharm Sin B. 2023;13(6):2559-71.
[https://doi.org/10.1016/j.apsb.2023.03.012]

-
Wu Y, Zhang F, Yang K, Fang S, Bu D, Li H, et al. SymMap: an integrative database of traditional Chinese medicine enhanced by symptom mapping. Nucleic Acids Res. 2019;47(D1):D1110-d7.
[https://doi.org/10.1093/nar/gky1021]

-
Fang S, Dong L, Liu L, Guo J, Zhao L, Zhang J, et al. HERB: a high-throughput experiment- and reference-guided database of traditional Chinese medicine. Nucleic Acids Res. 2021;49(D1):D1197-d206.
[https://doi.org/10.1093/nar/gkaa1063]

-
Yan D, Zheng G, Wang C, Chen Z, Mao T, Gao J, et al. HIT 2.0: an enhanced platform for Herbal Ingredients' Targets. Nucleic Acids Res. 2022;50(D1):D1238-d43.
[https://doi.org/10.1093/nar/gkab1011]

-
Liu X, Liu J, Fu B, Chen R, Jiang J, Chen H, et al. DCABM-TCM: A Database of Constituents Absorbed into the Blood and Metabolites of Traditional Chinese Medicine. J Chem Inf Model. 2023;63(15):4948-59.
[https://doi.org/10.1021/acs.jcim.3c00365]

-
Kim SK, Lee MK, Jang H, Lee JJ, Lee S, Jang Y, et al. TM-MC 2.0: an enhanced chemical database of medicinal materials in Northeast Asian traditional medicine. BMC Complement Med Ther. 2024;24(1):40.
[https://doi.org/10.1186/s12906-023-04331-y]

-
Wu W, Yang S, Liu P, Yin L, Gong Q, Zhu W. Systems Pharmacology-Based Strategy to Investigate Pharmacological Mechanisms of Radix Puerariae for Treatment of Hypertension. Front Pharmacol. 2020;11:345.
[https://doi.org/10.3389/fphar.2020.00345]

-
Liu H, Wang J, Zhou W, Wang Y, Yang L. Systems approaches and polypharmacology for drug discovery from herbal medicines: an example using licorice. J Ethnopharmacol. 2013;146(3):773-93.
[https://doi.org/10.1016/j.jep.2013.02.004]

-
Tao W, Xu X, Wang X, Li B, Wang Y, Li Y, et al. Network pharmacology-based prediction of the active ingredients and potential targets of Chinese herbal Radix Curcumae formula for application to cardiovascular disease. Journal of ethnopharmacology. 2013;145(1):1-10.
[https://doi.org/10.1016/j.jep.2012.09.051]

-
Zhang N-D, Han T, Huang B-K, Rahman K, Jiang Y-P, Xu H-T, et al. Traditional Chinese medicine formulas for the treatment of osteoporosis: implication for antiosteoporotic drug discovery. Journal of ethnopharmacology. 2016;189:61-80.
[https://doi.org/10.1016/j.jep.2016.05.025]

-
Zhang R, Zhu X, Bai H, Ning K. Network Pharmacology Databases for Traditional Chinese Medicine: Review and Assessment. Front Pharmacol. 2019;10:123.
[https://doi.org/10.3389/fphar.2019.00123]

-
Li X, Wei S, Niu S, Ma X, Li H, Jing M, et al. Network pharmacology prediction and molecular docking-based strategy to explore the potential mechanism of Huanglian Jiedu Decoction against sepsis. Comput Biol Med. 2022;144:105389.
[https://doi.org/10.1016/j.compbiomed.2022.105389]

-
Fang J, Wang L, Wu T, Yang C, Gao L, Cai H, et al. Network pharmacology-based study on the mechanism of action for herbal medicines in Alzheimer treatment. J Ethnopharmacol. 2017;196:281-92.
[https://doi.org/10.1016/j.jep.2016.11.034]

-
Wang X, Wu M, Lai X, Zheng J, Hu M, Li Y, et al. Network pharmacology to uncover the biological basis of spleen qi deficiency syndrome and herbal treatment. Oxidative medicine and cellular longevity. 2020;2020(1):2974268.
[https://doi.org/10.1155/2020/2974268]

-
Tran MN, Kim S, Nguyen QHN, Lee S. Molecular mechanisms underlying qi-invigorating effects in traditional medicine: Network pharmacology-based study on the unique functions of qi-invigorating herb group. Plants. 2022;11(19):2470.
[https://doi.org/10.3390/plants11192470]

-
Li S, Zhang B. Traditional Chinese medicine network pharmacology: theory, methodology and application. Chin J Nat Med. 2013;11(2):110-20.
[https://doi.org/10.1016/S1875-5364(13)60037-0]

-
Fan M, Jin C, Li D, Deng Y, Yao L, Chen Y, et al. Multi-level advances in databases related to systems pharmacology in traditional Chinese medicine: a 60-year review. Front Pharmacol. 2023;14:1289901.
[https://doi.org/10.3389/fphar.2023.1289901]

-
Lee M, Shin H, Park M, Kim A, Cha S, Lee H. Systems pharmacology approaches in herbal medicine research: a brief review. BMB Rep. 2022;55(9):417-28.
[https://doi.org/10.5483/BMBRep.2022.55.9.102]

-
Choi G, Kang Y, Moon B, Kim H. A comparative study about the origins of crude drugs in the northeast asian pharmacopoeias: centered on same name of materials but different genus. The Korea Journal of Herbology. 2013;28(5).
[https://doi.org/10.6116/kjh.2013.28.5.103]

