네트워크 약리학적 분석에 의한 소세포폐암에 대한 청대의 항암기전 연구
Ⓒ The Society of Pathology in Korean Medicine, The Physiological Society of Korean Medicine
Abstract
Lung cancer is the leading cause of cancer-related deaths worldwide. Indigo Naturalis (IN) is a dark blue powder obtained by processing leaves or stems of indigo plants, its anticancer effects have been reported in several studies. However, the pharmacological mechanism of IN in small cell lung cancer (SCLC) is not elucidated. In this study, to investigate the anticancer efficacy of IN for SCLC, we presented potential active ingredients, SCLC-related targets, and pharmacological mechanisms of IN that are expected to have anticancer activity for SCLC using a network pharmacological analysis. The phytochemical compounds of IN have been collected through TCMSP, SymMap, or HPLC documents. The active ingredients of IN such as indirubin, indican, isatin, and tryptanthrin were selected through ADME parameters or literature investigations for each compound. Using the Compounds, Disease-Target associations Databases, 124 common targets of IN and SCLC were obtained. Gene Ontology (GO), Kyoto Encyclopedia of Genes and Genomes (KEGG) Pathway enrichment analysis was carried out. GO biological processes are associated with response to xenobiotic stimulus, positive regulation of protein phosphorylation, regulation of mitotic cell cycle, and regulation of apoptotic signaling pathway. KEGG disease pathways included Gastric cancer, Bladder cancer, SCLC, and Melanoma. The main anticancer targets of the IN for SCLC were analyzed in 14 targets, including BCL2, MYC, and TP53. In conclusion, the results of this study based on the network pharmacology of IN can provide important data for the effective prevention and treatment of SCLC.
Keywords:
Indigo Naturalis, Small cell lung cancer, Anticancer mechanism, Network pharmacological analysis서 론
폐암은 전 세계적으로 유방암과 함께 가장 많이 진단되는 암 중 하나이며, 암 관련 사망의 주요 원인으로 알려져 있다. 2020년에 발생한 신규 암 환자 1,900만여 명 중 폐암 환자는 220만여 명이었으며, 암 관련 사망자 1,000만여 명 중 180만여 명이 폐암으로 인한 사망자로 집계되었다1). 그 중에서도 원발성 폐암의 약 15%를 차지하는 소세포폐암 (Small cell lung cancer, SCLC) 은 비소세포폐암 (Non-small cell lung cancer, NSCLC)에 비해 두 배 이상 빠른 증식, 광범위한 전이 양상, 다제내성 등의 특징을 보여 NSCLC에 비해 악성도가 높고 예후가 불량하다2,3). SCLC의 일반적인 치료법은 화학요법이며, 다른 가능한 치료법으로는 방사선 치료, 수술, 표적 치료, 면역 요법 등이 알려져 있다. 그러나 SCLC는 조기 진단이 어렵고 진행이 빨라 발견 시 대부분 수술적 처치가 어려우며, 초기 화학요법 후에도 재발률이 높아 이후 항암제에 대한 다제내성을 보이는 경우가 많다4,5). 또 대다수 암의 장기 생존율이 50여 년 전과 비교하여 SCLC의 장기 생존율은 거의 개선되지 않았다6). 지난 30년 간 SCLC의 치료법은 크게 변하지 않았으며, 현재까지 유효한 표적 치료법이 없어 새로운 치료법이 꾸준히 요구되고 있다7). 현재 SCLC의 5년 생존율은 약 7%에 불과하다8).
천연물은 일반적으로 다수의 화합물 포함하여 체내 활성 범위가 넓으며, 이를 병용 시 다양한 상승효과를 나타낸다고 알려져 있다9). 천연물은 특히 암과 관련된 다양한 경로에 작용하여 기존의 단일 표적 치료법보다 효과적일 수 있으며, 일부 천연물의 독특한 구조는 암 치료의 주된 문제인 약물 내성을 극복할 수 있는 가능성이 높다10). 한의학에서는 지난 수천 년 동안 암을 포함한 다양한 질병을 치료하기 위해 천연물을 사용해 왔으며, 최근 많은 전임상 연구에서 천연물에 대한 항암 효과가 보고되어 왔다. 대표적으로 雷公藤은 백혈병, 유방암, 간암 등에 대한 치료 효능이 알려졌으며11), 黃芩, 人蔘, 紫草, 黃連 등에 대한 다양한 항암 효능이 보고된 바 있다12). 천연물의 항암 작용기전이 밝혀진 taxol, vincristine, camptothecin, podophyllotoxin 등이 오랜 기간 효과적으로 사용되어 왔다는 사실은 암 치료에 대한 천연물 연구의 높은 잠재적 가치를 시사한다.
청대 (Indigo Naturalis, IN) 는 잎 또는 줄기를 건조, 발효 및 가공하여 얻는 암청색 분말로써 한의학에서는 淸熱劑로 주로 활용되고 있다13). 전통적으로 청대는 淸熱解毒, 涼血消斑, 瀉火定驚 등의 효능이 있으며, 다른 약재와 함께 배합되어 溫毒發斑, 血熱吐衄, 胸痛咳血, 口瘡, 痄腮, 喉痹, 小兒驚癇 과 같은 질환에 처방되어 왔다14). 최근 청대의 지혈, 해열, 항염, 진정, 항암 효능이 확인되었으며15), 특히 중국의 전통 처방 當歸龍薈丸 에서 만성 골수성 백혈병 의 주요한 치료 약물로 주목된 바 있다. 또한 다기관 임상 연구를 통해 급성 골수성 백혈병에 대한ATRA (All-trans-retinoic acid) - RIF (Realgar-indigo naturalis formula) 병용요법의 효능 및 안전성이 보고되었다16,17). 청대의 주요 활성 성분으로는 indirubin, tryptanthrin이 알려져 있다18). Indirubin은 다수의 세포 및 동물실험에서 JAK/STAT3, CDK 신호전달 경로 등을 억제하는 기전을 통해 항증식, 항혈관신생, 항암 효능을 지닌다고 보고되었으며, 최근에는 건선, 궤양성 대장염 등 자가면역질환에 관련한 면역조절 효과가 활발히 연구되고 있다19-24). Tryptanthrin 또한 항암, 항염증, 항원충, 항알레르기, 항산화, 항진균, 항균 효능 등이 알려져 있다25). 위와 같은 청대의 다양한 효능 보고에도 불구하고 청대의 다양한 활성 성분과 관련 타깃 유전자의 상호작용에 대한 연구는 거의 이루어진 바 없다. 네트워크 약리학은 비교적 최근에 나타나 활발히 연구되고 있는 분야로, 약리학적 데이터에 네트워크 분석 기법을 적용하여 특정 약물이 분자 수준에서 생물학적 시스템에 미치는 영향을 설명하는 분야이다. 네트워크 약리학은 체내에서 상호작용하는 다수의 단백질과 약물 사이의 관계를 네트워크로 표현할 수 있음을 기본 전제로, 궁극적으로는 이를 활용한 보다 효과적이고 안전한 약물 개발을 목표로 하고 있다. 네트워크 약리학을 활용하여 특정 약물의 기존 효과뿐만 아니라 새로운 적응증을 보다 효율적으로 식별할 수 있으므로, 이는 특히 다양한 종류의 활성 성분으로 이루어진 천연물, 또는 한약의 효능 및 작용 기전을 탐색하는 데 유용한 연구 방법이다.
본 연구에서는 청대의 네트워크 약리학적 분석 방법을 활용하여 SCLC에 항암 활성이 기대되는 주요 활성 성분을 다수 선별하여, 이와 관련된 타깃 단백질과 핵심 작용기전을 규명하여 SCLC의 예방과 효율적인 치료를 위한 생물정보학적 기초자료를 제공하고자 하였다.
재료 및 방법
1. Identification of Active compounds in IN
TCMSP (https://tcmsp-e.com/index.php), SymMap (http://www.symmap.org/) Databases를 이용하여 청대 구성 화합물을 수집하였으며, 관련 정량 분석 문헌을 참고하여 구성 화합물을 추가 수집하였다. TCMSP는 한약재의 성분, 표적, 질병 간 상관관계에 대한 정보와 함께 이에 대한 시스템 약리학적 분석 결과를 제공하며, SymMap 또한 전통 중의학과 서양의학에 근거한 방대한 정보를 유사한 방식으로 제공하고 있다26,27).
각 화합물의 약물 유사성 (Drug likeness, DL), 5 법칙 (Lipinski’s Rule of 5, RO5) 충족 여부, 장내 흡수도 (GI absorption)를 예측하기 위해 SwissADME(http://www.swissadme.ch) Database를 이용하였으며, 해당 Database의 평가 기준을 활용하여 청대 활성 성분을 선정하였다.
2. Screening of Targets related to Active compounds in IN and SCLC
UniProt (https://www.uniprot.org/), CTD (http://ctdbase.org/) Databases를 이용하여 각 활성 성분에 대한 타깃을 확보하였으며, 이를 청대 관련 타깃으로 선정하였다. 연구 과정에서 수집한 모든 gene ID는 UniProt ID로 변환하여 표준화하였다.
3. Functional enrichment analysis for IN-SCLC Common targets
SCLC의 공통 타깃에 대한 Gene ontology (GO), Kyoto Encyclopedia of Genes and Genomes (KEGG) enrichment analysis를 Database for Annotation, Visualization and Integrated Discovery (DAVID) 6.8 Gene Functional Classification Tool (https://david.ncifcrf.gov/)과 KEGG website (https://www.genome.jp /kegg/)에서 시행하였다.
4. PPI network construction and Screening of hub targets
Cytoscape 3.9.1, Search Tool for the Retrieval of Interacting Genes (STRING) 11.5 Database를 통해 공통 타깃에 대한 Protein-protein interaction (PPI) 네트워크를 구성하였다.
5. Identification of Core targets involving in SCLC pathway
KEGG Mapper (https://www.genome.jp/kegg/mapper/) 를 이용하여 SCLC pathway에 포함된 청대 활성 성분의 핵심 타깃을 확인하였다.
결 과
1. Active compounds in IN
TCMSP, SymMap 을 이용하여 청대 구성 화합물 69종을 확보하였으며, 검색어로는 “Indigo Naturalis”, “Qing Dai”를 사용하였다. 관련 HPLC 문헌28,29)을 참고하여 청대 구성 화합물 63종을 구성 목록에 추가하였으며, 중복되거나 구조식을 확인할 수 없는 화합물을 제외하여 청대 구성 화합물 108종을 최종 선정하였다.
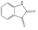
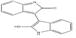
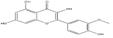
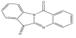
선정된 청대 화합물의 구조식은 Pubchem (https://www.ncbi.nlm.nih.gov), Open Babel (http://openbabel.org) 에서 canonical SMILES로 변환되었으며, SwissADME에서 제공하는 Bioavailability Radar Plot을 통해 DL 기준을 만족하는 3개의 활성 성분(Indican, Isolariciresinol, Lariciresinol) 을 선별하였다. 이외에 위의 기준을 만족하지는 못하였으나, 체내 약리 활성이 비교적 잘 알려진 Indirubin, Tryptanthrin, Isatin 등의 화합물 13종을 청대의 활성 성분에 포함하였다(Table 1).
2. Targets related to Active compounds in IN
청대 활성 성분에 관련된 타깃을 조사하기 위해 UniProt, CTD를 이용하였으며, 청대 관련 타깃 262개를 확보하였다.
3. IN related Targets for SCLC treatment
DisGeNET, GeneCards, CTD를 통해 SCLC 관련 타깃을 각 885개, 18,295개, 14,232개 수집하였으며, 이후 DisGeNET, GeneCards, CTD에서 삼중으로 수집된 SCLC 관련 타깃 786개와 청대 관련 타깃 262개의 교집합을 분석하였으며, 이를 통해 청대와 SCLC의 공통 타깃으로 타깃 124개를 선정하였다(Table 2).
4. GO and KEGG pathway enrichment analysis
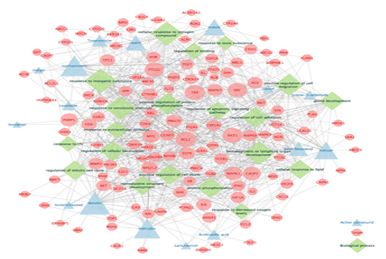
공통 타깃 124개가 관여하는 주요 생물학적 경로와 질병 기전을 탐색하기 위해 Enrichment analysis를 시행하였다. 분석 결과, Cellular components (CC) 에서 98개, Molecular functions (MF) 에서 151개, Biological processes (BP) 에서 1,753개, KEGG pathways에서 190개의 세부 항목을 얻었다. 유의성에 따른 GO 상위 10개, KEGG pathways 상위 20개의 분석 결과를 도식화 하였다(Fig. 1).
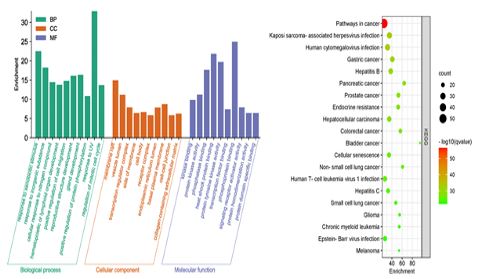
Enrichment Analysis of GO and KEGG Pathway for 124 Common Targets. (A) GO enrichment analysis. (B) KEGG enrichment analysis.
CC에는 “membrane raft”, “vesicle lumen”, “transcription regulator complex”, MF에는 “kinase binding”, “protein kinase activity”, “heat shock protein binding”, “protein tyrosine kinase activity”, “transcription factor binding”, “MAP kinase activity” 등이 포함되었으며, BP에는 주로 “response to xenobiotic stimulus”, “positive regulation of protein phosphorylation”, “regulation of mitotic cell cycle”, “regulation of apoptotic signaling pathway” 등이 관련 경로로 포함되었다. KEGG pathways에는 “Pathways in cancer”, “Gastric cancer”, “Bladder cancer”, “Small cell lung cancer”, “Melanoma” 등이 포함되었다.
5. Construction of C-T-BP Network
Cytoscape 3.9.1을 이용하여, 청대 활성 성분 16종, 공통 타깃 124개, Biological process 상위 20개에 대한 Compound-Target-Biological process (C-T-BP) 네트워크를 구성하였다(Fig. 2).
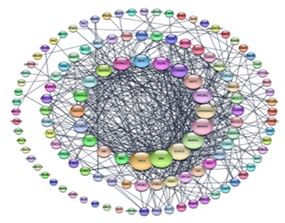
6. PPI network construction and Screening of hub targets and protein modules
STRING Database를 이용하여 PPI 네트워크를 구성하여 TP53, CDKN1A, CDK4, CDK2, CCND1, RB1, CDKN1B, E2F1, SRC, MAPK3 등의 순서상호연결성이 높은 허브 타깃 20개를 탐색하였다(Fig. 3).
7. Identification of IN-SCLC Core targets involving in SCLC pathway
허브 타깃 상위 20개를 SCLC에 대한 청대 활성 성분의 핵심 타깃으로 선정하여 핵심 타깃이 관여하는 SCLC 신호전달 경로를 확인하기 위해 KEGG Mapper를 이용하였으며, AKT1, BCL2, CASP3, CCND1, CDK2, CDK4, CDKN1A, CDKN1B, E2F1, ITGB1, MYC, PIK3R1, RB1, TP53이 관련된 유전자로 분석되었다 (Table 3).
고 찰
본 연구에서는 TCMSP, SymMap, SwissADME, STP, SEA, UniProt, DisGeNet 등 최근 발전한 생물정보학적 데이터베이스와 일련의 네트워크 약리학적 분석 방법을 활용하여SCLC에 주요한 항암 효과가 기대되는 청대의 활성 성분, 관련 타깃 및 기전을 확인하였다.
분석 결과, 청대 활성 성분 16종이 선별되었으며, 청대 활성 성분과 SCLC 타깃은 Betulin 36개, Isorhamnetin 31개, Indirubin 29개 등의 순서로 관련되었다 (Fig. 2). 청대 타깃이 관여하는 BP에는 주로 무기물에 대한 반응, 단백질 인산화 양성 조절, 체세포 분열주기 조절, 세포사멸 신호전달 경로 조절 등이 포함되었으며, KEGG에는 위암, 방광암, 소세포폐암, 흑색종 등이 포함되었다. 핵심 타깃의 질병 경로는 KEGG Mapper를 통해 조사되었으며, beta-sitosterol, betulin, indican, indirubin, indole, isatin, isolariciresinol, isorhamnetin, tryptanthrin 등이 SCLC에 대한 주요한 활성 성분으로 분석되었다.
청대의 잘 알려진 지표성분으로는 indirubin, indigo가 중국약전에 소개되어 있다14). Indirubin은 인디고 식물 (Indigo plant)과 일부 동물에서 생성되는 indole alkaloid로, 다양한 인간 암 세포주에서 항암 활성을 나타냈으며, 항염증, 면역 조절, 항산화 효능 또한 보고되어 있다11). 현재, 정확한 항암 작용기전이 밝혀지고 있으며, 특히 indirubin은 암과 관련하여 JAK/STAT3, Wnt/β-Catenin 신호전달 경로의 활성을 조절하며, CDK2의 저해제로 잘 알려져 있다20,30,31). Isatin은 많은 식물에서 발견되는 indole 유도체로 유방, 폐, 결장을 포함한 다양한 암 세포의 성장을 억제하는 것으로 알려져 있다. 또한, 이 compound는 항알레르기, 항말라리아, 항바이러스, 항균 효과 등이 있으며, CASP3을 활성화하거나 CDK2와 CDKN1A를 억제함으로써 세포사멸을 유도할 수 있다고 보고되었다32-34). 특히, Tryptanthrin은 Indigo를 승화하는 과정에서 처음 발견된 indoloquinazoline alkaloid로, 이후 균류 및 다양한 식물에서 분리되었다. 또한, 이 compound는 항균, 항진균, 항원충 및 항기생충 활성, COX2, 5LOX, NO 합성효소 및 PGE2 발현 억제, 세포독성 및 항암 효능이 보고되어 있다35).
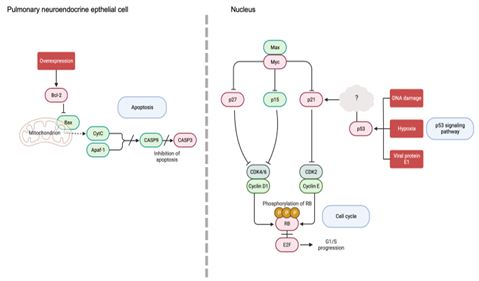
KEGG Mapper를 통해 SCLC 신호전달 경로에 포함된 청대 핵심 타깃은 AKT1, BCL2, CASP3, CCND1, CDK2, CDK4, CDKN1A, CDKN1B, E2F1, ITGB1, MYC, PIK3R1, RB1, TP53로 확인되었다. 분석된 타깃 중 SCLC에서 과발현되는 주요 항암 표적인자로 bcl-2, c-myc가 알려져 있다36). Bcl-2는 SCLC를 포함한 많은 종류의 암에서 과발현되는 항세포사멸 단백질로, 과잉 발현되면 암세포가 생존할 가능성이 높다. Bcl-2는 미토콘드리아 막 투과성을 조절하여 세포사멸을 조절하는데, 미토콘드리아로부터 cytochrome c의 방출을 방지하거나APAF1에 결합함으로써 caspase 활성을 억제하며, C-myc는 세포주기 진행과 세포분열에 관여하는 유전자의 발현을 상향조절함으로써 암세포의 성장과 증식을 촉진한다37). 그러나 현재 Bcl-2를 표적으로 시행된 임상 2상 연구에서 표적 항암제 Oblimersen, Obatoclax이 최종 탈락하였으며, C-myc를 단일 타깃으로 하는 표적 항암제는 현재까지 개발되지 못하였다36,38). 암억제단백질인p53은 세포주기 및 세포사멸을 조절하는 전사 인자이다39). 절반 이상의 암 환자에게서 p53 변이가 관찰되며, 특히 SCLC 환자의 약 80% 이상에서 주로 p53의 기능 손실 (loss-of-function) 이 보고되었다7,40). 암 억제와 관련한 p53 신호전달 경로의 주요 하부 인자로 P21은 CDK의 활성을 억제하는 CDK 저해제로 주요 기질을 인산화하고 세포주기 진행을 막는다고 알려져 있다41,42). P21은 p53에 독립적인 경로를 통해서도 유도될 수 있는데, 예를 들어 Peng H 등의 연구에서는 p53이 손실된 만성 골수성 백혈병 세포주 K562 세포주에서 indirubin 유도체가 time-dose-dependent하게 p21을 상향 조절하였으며, 암세포의 성장이 저해되었다43). 이는 대부분 p53 유전자의 기능 손실을 가지는 SCLC에서 indirubin이 p21에 p53-independent하게 작용할 수 있을 것으로 예상한다. 예를 들어, indirubin은 p21을 상향조절하고, 암 세포의 CDK2 활성을 억제하여 RB 인산화를 저해할 수 있으며, 이는 G1-S transition에서 세포주기를 차단하여 SCLC 세포사멸로 이어질 수 있다 (Fig. 4).
본 연구는 네트워크 약리학적 분석을 통해 청대의 잠재적 활성 성분 및 관련 타깃을 확보하였으며, 특히 indirubin을 중심으로 SCLC에 대한 청대의 항암 효능을 분석 및 예측하였다. 앞으로 본 연구결과를 기반으로 실험적인 검증을 통하여 전통의학에서 검증된 한약재에 대한 과학적인 근거를 제시할 수 있을 것이다.
결 론
소세포폐암에 대한 청대 주요 활성 성분으로 indirubin, isatin, tryptanthrin 등이 선별되었으며, 주요 항암 표적인자로는 AKT1, BCL2, CDK2, CASP3, E2F1, TP53 등이 포함되었다. 청대는 세포주기, 세포사멸 신호전달 경로 등에 관여하여 암세포의 증식, 세포사멸 및 혈관신생을 조절할 수 있으며, 이를 통해 SCLC에 대한 항암 효과를 나타낼 수 있다. 본 연구는 네트워크 약리학을 기반으로 청대 활성성분의 잠재적 작용기전과 효과적인 SCLC 치료법의 발전으로 이어질 수 있는 청대의 생물정보학적 기초자료를 제공하였으며, 향후 효율적인 실험 설계를 위한 이론적 근거를 제시하였다.
Acknowledgments
본 연구는 부산대학교 기본연구지원사업(2년)의 지원을 받아 수행되었음.
References
- International Agency for Research on Cancer. Global Cancer Observatory: Cancer Today. Available from: https://gco.iarc.fr/today/fact-sheets-cancers, [Accessed 28th September 2022].
-
Byers LA, Rudin CM. Small cell lung cancer: where do we go from here? Cancer. 2015;121(5):664-72.
[https://doi.org/10.1002/cncr.29098]

-
Herzog BH, Devarakonda S, Govindan R. Overcoming Chemotherapy Resistance in SCLC. J Thorac Oncol. 2021;16(12):2002-15.
[https://doi.org/10.1016/j.jtho.2021.07.018]

-
Rudin CM, Brambilla E, Faivre-Finn C, Sage J. Small-cell lung cancer. Nat Rev Dis Primers. 2021;7(1):1-20.
[https://doi.org/10.1038/s41572-020-00235-0]

-
Minami T, Kijima T, Kohmo S, Arase H, Otani Y, Nagatomo I, et al. Overcoming chemoresistance of small-cell lung cancer through stepwise HER2-targeted antibody-dependent cell-mediated cytotoxicity and VEGF-targeted antiangiogenesis. Sci Rep. 2013;3:2669:1-11.
[https://doi.org/10.1038/srep02669]

-
Quaresma M, Coleman MP, Rachet B. 40-year trends in an index of survival for all cancers combined and survival adjusted for age and sex for each cancer in England and Wales, 1971-2011: a population-based study. Lancet. 2015;385(9974):1206-18.
[https://doi.org/10.1016/S0140-6736(14)61396-9]

-
Karachaliou N, Pilotto S, Lazzari C, Bria E, de Marinis F, Rosell R. Cellular and molecular biology of small cell lung cancer: an overview. Transl Lung Cancer Res. 2016;5(1):2-15.
[https://doi.org/10.21037/tcr.2016.04.04]

- Cancer.Net Editorial Board. Lung cancer - small cell - statistics. Available from: https://www.cancer.net/cancer-types/lung-cancer-small-cell/statistics, [Accessed 28th September 2022].
-
Pezzani R, Salehi B, Vitalini S, Iriti M, Zuniga FA, Sharifi-Rad J, et al. Synergistic Effects of Plant Derivatives and Conventional Chemotherapeutic Agents: An Update on the Cancer Perspective. Medicina (Kaunas). 2019;55(4):1-16.
[https://doi.org/10.3390/medicina55040110]

-
Castaneda AM, Melendez CM, Uribe D, Pedroza-Diaz J. Synergistic effects of natural compounds and conventional chemotherapeutic agents: recent insights for the development of cancer treatment strategies. Heliyon. 2022;8(6):e09519.
[https://doi.org/10.1016/j.heliyon.2022.e09519]

-
Liu Z, Ma L, Zhou GB. The main anticancer bullets of the Chinese medicinal herb, thunder god vine. Molecules. 2011;16(6):5283-97.
[https://doi.org/10.3390/molecules16065283]

-
Tan W, Lu J, Huang M, Li Y, Chen M, Wu G, et al. Anti-cancer natural products isolated from chinese medicinal herbs. Chin Med. 2011;6(1):27:1-15.
[https://doi.org/10.1186/1749-8546-6-27]

- Stasiak N, Kukula-Koch W, Glowniak K. Modern industrial and pharmacological applications of indigo dye and its derivatives—a review. Acta Pol Pharm. 2014;71(2):215-21.
- State Pharmacopoeia Commission of the PRC. Pharmacopoeia of the People's Republic of China. China: People's Medical Publishing House; 2005.
-
Tang W., Eisenbrand G. Chinese Drugs of Plant Origin: Chemistry, Pharmacology, and Use in Traditional and Modern Medicine. Berlin: Springer-Verlag; 1992.
[https://doi.org/10.1007/978-3-642-73739-8]

-
Han J. Traditional Chinese medicine and the search for new antineoplastic drugs. J Ethnopharmacol. 1988;24(1):1-17.
[https://doi.org/10.1016/0378-8741(88)90135-3]

-
Yang MH, Wan WQ, Luo JS, Zheng MC, Huang K, Yang LH, et al. Multicenter randomized trial of arsenic trioxide and Realgar-Indigo naturalis formula in pediatric patients with acute promyelocytic leukemia: Interim results of the SCCLG-APL clinical study. Am J Hematol. 2018;93(12):1467-73.
[https://doi.org/10.1002/ajh.25271]

-
Liau BC, Jong TT, Lee MR, Chen SS. LC-APCI-MS method for detection and analysis of tryptanthrin, indigo, and indirubin in daqingye and banlangen. J Pharm Biomed Anal. 2007;43(1):346-51.
[https://doi.org/10.1016/j.jpba.2006.06.029]

-
Cao Z, Yang F, Wang J, Gu Z, Lin S, Wang P, et al. Indirubin Derivatives as Dual Inhibitors Targeting Cyclin-Dependent Kinase and Histone Deacetylase for Treating Cancer. J Med Chem. 2021;64(20):15280-96.
[https://doi.org/10.1021/acs.jmedchem.1c01311]

-
Nam S, Buettner R, Turkson J, Kim D, Cheng JQ, Muehlbeyer S, et al. Indirubin derivatives inhibit Stat3 signaling and induce apoptosis in human cancer cells. Proc Natl Acad Sci U S A. 2005;102(17):5998-6003.
[https://doi.org/10.1073/pnas.0409467102]

-
Wang H, Wang Z, Wei C, Wang J, Xu Y, Bai G, et al. Anticancer potential of indirubins in medicinal chemistry: Biological activity, structural modification, and structure-activity relationship. Eur J Med Chem. 2021;223:113652:1-30.
[https://doi.org/10.1016/j.ejmech.2021.113652]

-
Hsieh WL, Lin YK, Tsai CN, Wang TM, Chen TY, Pang JH. Indirubin, an acting component of indigo naturalis, inhibits EGFR activation and EGF-induced CDC25B gene expression in epidermal keratinocytes. J Dermatol Sci. 2012;67(2):140-6.
[https://doi.org/10.1016/j.jdermsci.2012.05.008]

-
Gao W, Guo Y, Wang C, Lin Y, Yu L, Sheng T, et al. Indirubin ameliorates dextran sulfate sodium-induced ulcerative colitis in mice through the inhibition of inflammation and the induction of Foxp3-expressing regulatory T cells. Acta Histochem. 2016;118(6):606-14.
[https://doi.org/10.1016/j.acthis.2016.06.004]

-
Lin YK, Chang SH, Yang CY, See LC, Lee BH, Shih IH. Efficacy and safety of indigo naturalis ointment in Treating Atopic Dermatitis: A randomized clinical trial. J Ethnopharmacol. 2020;250:112477:1-8.
[https://doi.org/10.1016/j.jep.2019.112477]

-
Kaur R, Manjal SK, Rawal RK, Kumar K. Recent synthetic and medicinal perspectives of tryptanthrin. Bioorg Med Chem. 2017;25(17):4533-52.
[https://doi.org/10.1016/j.bmc.2017.07.003]

-
Chiang YR, Li A, Leu YL, Fang JY, Lin YK. An in vitro study of the antimicrobial effects of indigo naturalis prepared from Strobilanthes formosanus Moore. Molecules. 2013;18(11):14381-96.
[https://doi.org/10.3390/molecules181114381]

-
Ru J, Li P, Wang J, Zhou W, Li B, Huang C, et al. TCMSP: a database of systems pharmacology for drug discovery from herbal medicines. J Cheminform. 2014;6:13:1-6.
[https://doi.org/10.1186/1758-2946-6-13]

-
Daina A, Michielin O, Zoete V. SwissADME: a free web tool to evaluate pharmacokinetics, drug-likeness and medicinal chemistry friendliness of small molecules. Sci Rep. 2017;7:42717:1-13.
[https://doi.org/10.1038/srep42717]

-
Zhou Y, Zhou B, Pache L, Chang M, Khodabakhshi AH, Tanaseichuk O, et al. Metascape provides a biologist-oriented resource for the analysis of systems-level datasets. Nat Commun. 2019;10(1):1523:1-10.
[https://doi.org/10.1038/s41467-019-09234-6]

-
Liu SG, Luo GP, Qu YB, Chen YF. Indirubin inhibits Wnt/beta-catenin signal pathway via promoter demethylation of WIF-1. BMC Complement Med Ther. 2020;20(1):250:1-10.
[https://doi.org/10.1186/s12906-020-03045-9]

-
Hoessel R, Leclerc S, Endicott JA, Nobel ME, Lawrie A, Tunnah P, et al. Indirubin, the active constituent of a Chinese antileukaemia medicine, inhibits cyclin-dependent kinases. Nat Cell Biol. 1999;1(1):60-7.
[https://doi.org/10.1038/9035]

- Premanathan M, Radhakrishnan S, Kulangiappar K, Singaravelu G, Thirumalaiarasu V, Sivakumar T, et al. Antioxidant & anticancer activities of isatin (1H-indole-2,3-dione), isolated from the flowers of Couroupita guianensis Aubl. Indian J Med Res. 2012;136(5):822-6.
-
Chen DL, Zhou D, Chu W, Herrbrich P, Engle JT, Griffin E, et al. Radiolabeled isatin binding to caspase-3 activation induced by anti-Fas antibody. Nucl Med Biol. 2012;39(1):137-44.
[https://doi.org/10.1016/j.nucmedbio.2011.08.001]

-
Ferraz de Paiva RE, Vieira EG, Rodrigues da Silva D, Wegermann CA, Costa Ferreira AM. Anticancer Compounds Based on Isatin-Derivatives: Strategies to Ameliorate Selectivity and Efficiency. Front Mol Biosci. 2020;7:627272:1-24.
[https://doi.org/10.3389/fmolb.2020.627272]

-
Jahng Y. Progress in the studies on tryptanthrin, an alkaloid of history. Arch Pharm Res. 2013;36(5):517-35.
[https://doi.org/10.1007/s12272-013-0091-9]

-
Yuan M, Zhao Y, Arkenau HT, Lao T, Chu L, Xu Q. Signal pathways and precision therapy of small-cell lung cancer. Signal Transduct Target Ther. 2022;7(1):187:1-18.
[https://doi.org/10.1038/s41392-022-01013-y]

-
Tsujimoto Y, Finger LR, Yunis J, Nowell PC, Croce CM. Cloning of the chromosome breakpoint of neoplastic B cells with the t(14;18) chromosome translocation. Science. 1984;226(4678):1097-9.
[https://doi.org/10.1126/science.6093263]

-
Wang C, Zhang J, Yin J, Gan Y, Xu S, Gu Y, et al. Alternative approaches to target Myc for cancer treatment. Signal Transduct Target Ther. 2021;6(1):117:1-14.
[https://doi.org/10.1038/s41392-021-00500-y]

-
Baugh EH, Ke H, Levine AJ, Bonneau RA, Chan CS. Why are there hotspot mutations in the TP53 gene in human cancers? Cell Death Differ. 2018;25(1):154-60.
[https://doi.org/10.1038/cdd.2017.180]

-
George J, Lim JS, Jang SJ, Cun Y, Ozretic L, Kong G, et al. Comprehensive genomic profiles of small cell lung cancer. Nature. 2015;524(7563):47-53.
[https://doi.org/10.1038/nature14664]

-
el-Deiry WS, Tokino T, Velculescu VE, Levy DB, Parsons R, Trent JM, et al. WAF1, a potential mediator of p53 tumor suppression. Cell. 1993;75(4):817-25.
[https://doi.org/10.1016/0092-8674(93)90500-P]

-
Karimian A, Ahmadi Y, Yousefi B. Multiple functions of p21 in cell cycle, apoptosis and transcriptional regulation after DNA damage. DNA Repair (Amst). 2016;42:63-71.
[https://doi.org/10.1016/j.dnarep.2016.04.008]

-
Peng H, Yuan X, Shi R, Wei X, Ren S, Yan C, et al. PHII-7 inhibits cell growth and induces apoptosis in leukemia cell line K562 as well as its MDR- counterpart K562/A02 through producing reactive oxygen species. Eur J Pharmacol. 2013;718(1-3):459-68.
[https://doi.org/10.1016/j.ejphar.2013.07.038]